- Pathologie /
- Forschung /
- Aktuelle Projekte
Aktuelle Projekte
Jüngste Erkenntnisse aus der Genomsequenzierung unterstreichen den wirtsspezifischen Charakter des Pilzes Pneumocystis und geben Aufschluss über mögliche Anforderungen an die Zellkultur, deren Erfolg bisher begrenzt war. Entscheidend dürfte die Verwendung von respiratorischem Gewebe des entsprechenden Wirtes sein. Diese Studie soll folgende Fragen beantworten: Zeigt Pneumocystis f. sp. suis (P. suis) eine strikte Wirtsspezifität zum und eine starke Nahrungsabhängigkeit vom porzinen Wirtsgewebe? Ist der Pilz nur für geringgradige Gewebsläsionen verantwortlich?
In Vorversuchen haben wir verschiedene Ansätze zur Kultivierung von Atemwegsgewebe des Schweines getestet. Es wurden Trachealexplantate und Monolayer aus Lungen-/Atemwegszellen vom Schwein kultiviert und letztere in eine Air/Liquid Interphase-Kultur überführt. In unserer Studie werden diese Systeme mit P. suis inokuliert. Für den Fall, dass die Anzucht nicht gelingt, wird parallel dazu ein zellfreies Medium-Kultivierungssystem etabliert, mit dem weitere Projektteile bearbeitet werden können. Für die Inokulation werden P. suis-Organismen entweder aus Bronchiallavageflüssigkeit oder den Lungen von immunsupprimierten Schweinen separiert, konzentriert und mittels Durchflusszytometrie unter Verwendung kommerziell erhältlicher Antikörper quantifiziert. Darüber hinaus werden monoklonale Anti-P. suis-Antikörper mit Hilfe der Hybridoma-Technologie hergestellt. Die P. suis-Stadien und die durch die Infektion entstandenen Gewebsläsionen werden durch Transmissionselektronenmikroskopie dargestellt. Schließlich wird die Genexpression von P. suis mit Hilfe von RNA-Seq untersucht.
P. suis ist eine der rudimentär beschriebenen Pneumocystis-Arten. Zellkulturen waren bisher nur begrenzt erfolgreich, und jeder Beitrag zu diesem Thema ist für die gesamte Pneumocystis-Gemeinschaft von wesentlicher Bedeutung. Erste Schritte zur Erzeugung monoklonaler Anti-P.-suis-Antikörper durch die Hybridoma-Technologie und deren Anwendung bei der Zellsortierung werden durchgeführt, um die Grundlage für künftige Projekte zu schaffen. Die bildgebende Darstellung wird das Verständnis der Pathogenese von P. suis fördern. Die Genexpressionsanalyse wird grundlegende biologische Funktionen aufdecken und zum Verständnis der Komplexität der Pilz-Wirt-Interaktionen beitragen.
Mitarbeiter:innen:
Priv. Doz. Dr. Christiane Weissenbacher-Lang
Dr. Barbara Blasi
Mag. Nicolle Gobbo Oliveira Erünlü
Univ. Prof. Dr. Herbert Weissenböck
Kooperationspartner:innen:
Prof. Dr. Ingrid Walter, Arbeitsgruppe für Histologie, Institut für Morphologie, Veterinärmedizinische Universität Wien
Mag. Natascha Leitner, Arbeitsgruppe für Histologie, Institut für Morphologie, Veterinärmedizinische Universität Wien
Kerstin Mair PhD, Institut für Immunologie, Veterinärmedizinische Universität Wien
Prof. Dr Andrea Ladinig, Universitätsklinik für Schweine, Veterinärmedizinische Universität Wien
Mag. Christian Knecht, Universitätsklinik für Schweine, Veterinärmedizinische Universität Wien
Dr. Joseph A. Kovacs, Critical Care Medicine Department, NIH Clinical Center, Bethesda, Maryland, USA
Dr. Liang Ma, Critical Care Medicine Department, NIH Clinical Center, Bethesda, Maryland, USA
Dr. Ousmane H. Cissé, Critical Care Medicine Department, NIH Clinical Center, Bethesda, Maryland, USA
Lisa R. Bishop, Critical Care Medicine Department, NIH Clinical Center, Bethesda, Maryland, USA
Dr Shelly J. Curran, Critical Care Medicine Department, NIH Clinical Center, Bethesda, Maryland, USA
Projektzeitraum: 03/2023 – 02/2027
Finanzierung:
FWF – Fonds zur Förderung der Wissenschaftlichen Forschung
Trotz intensiver Forschung bleibt Herzstillstand eine der häufigsten Todesursachen der modernen Gesellschaft. Nur sehr wenige Patienten überleben einen Herzstillstand mit gutem neurologischem Outcome. Am Tiermodell (Ratte und Schwein) werden die Auswirkungen von Herzstillstand und Reperfusion auf das Gehirn untersucht. Als therapeutische Ansätze werden unterschiedliche Reanimationsarten und milde Hypothermie getestet.
Beteiligte Mitarbeiter:
Dr. Sandra Högler
Univ.-Prof. Dr. Peter Schmidt, Dipl.ECVP
Kooperationspartner:
Dr. Sandra Högler, Abteilung für Labortierpathologie, Vetmeduni Vienna
Prof. Sterz, Dr. Wolfgang Weihs, Department für Notfallmedizin, Med.Uni.Vienna
Prof. Sipos, Klinik für Schweine, Vetmeduni Vienna
Catharina Duvigneau, Institut für Biochemie, Vetmeduni Vienna
Finanzierung:
FWF - Fonds zur Förderung der Wissenschaftlichen Forschung
Hochschuljubiläumsstiftung der Stadt Wien
AKH
Im Rahmen der Diagnostik übermittelte tote Tiere stellen häufig das Initialereignis bei der Erkennung und weiteren Nachverfolgung neuartiger Krankheiten dar (Beispiele: Usutu Virus Infektion, West Nile Virus Infektion, Rustrela Virus Infektion).
Darauf aufbauend werden in manchen Fällen Überwachungs- und Monitoringprogramme initiiert, im Rahmen welcher Daten zur Inzidenz, Epidemiologie und generellen Bedeutung neu auftretender Krankheiten erhoben werden.
Beteiligte Mitarbeiter:
Univ.-Prof. Dr. Herbert Weissenböck, Dipl.ECPHM
Priv.-Doz. Dr. Christiane Weissenbacher-Lang
Kooperationspartner:
Institut für Virologie
Universitätsklinik für Schweine
AGES
Friedrich-Loeffler-Institut, Greifswald - Insel Riems
Klinische und vergleichende Neuropathologie, Institut für Tierpathologie, LMU München
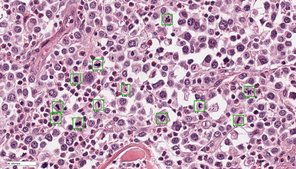
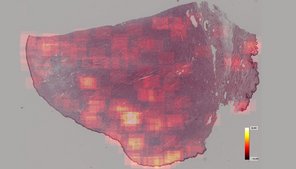
Während in der Pathologie traditionell Gewebeproben mithilfe eines Lichtmikroskops untersucht werden, setzt die digitale Pathologie auf das Erstellen, Verwalten und Untersuchen von digitalen Mikroskopiebildern. Sogenannte Whole-Slide Image (WSI) Scanner ermöglichen das Digitalisieren von gesamten histologischen Präparaten, welche anschließend digital durch UntersucherInnen betrachtet (digitale Mikroskopie) oder durch Computersoftware ausgewertet (automatisierte Bildanalyse) werden können. Es ergeben sich zahlreiche innovative Anwendungsmöglichkeiten für die histopathologische Diagnostik, Forschung und Lehre. Hinsichtlich der Diagnostik von chirurgischen Gewebeproben sind generelle Vorteile der digitalen Pathologie, im Vergleich zur herkömmlichen Lichtmikroskopie, unter anderem die hohe Arbeitsflexibilität (z.B Homeoffice), Optimierung der Arbeitsabflüsse und das genauere und reproduzierbarere Bestimmen von quantitativen Parametern. Letztere kann vor allem durch automatisierte Bildanalysen, insbesondere mittels Methoden der künstlichen Intelligenz, erreicht werden. Obwohl diagnostische Labore die digitale Pathologie zunehmend als Untersuchungsmodalität integrieren, werden zurzeit automatisierte Bildanalysen allerdings aufgrund der geringen Verfügbarkeit von Bildanalysesoftware und der unzureichenden Erkenntnisse über die Leistung und Zuverlässigkeit noch nicht routinemäßig angewendet.
Die Zielsetzung dieses Forschungsprojektes ist es innovative Softwarelösungen unter Einbeziehung von Methoden der künstlichen Intelligenz zu entwickeln und für die diagnostische Anwendung zu validieren. Zusammen mit Kooperationspartners von der Technischen Hochschule Ingolstadt (Arbeitsgruppe Prof. Dr. Marc Aubreville), Friedrich-Alexander Universität Erlangen-Nürnberg (Arbeitsgruppe Prof. Dr. Katharina Breininger), Freien Universität Berlin (Arbeitsgruppe Prof. Dr. Robert Klopfleisch) und Animal Medical Center New York (Dr. Taryn A Donovan) werden verschiedene relevante diagnostische Fragestellungen, insbesondere auf dem Gebiet der Tumorpathologie, bearbeitet. Schwerpunkt sind die Optimierung der Algorithmen, um eine breite Anwendung in verschiedenen Laboren zu ermöglichen, die verschiedenen Anwendungsmöglichkeiten zu erforschen, den Einfluss auf Qualität und Effizienz der Diagnose / Prognose zu untersuchen sowie Vor- und Nachteile bzw. Limitationen der automatisierten Bildanalyse herauszuarbeiten.