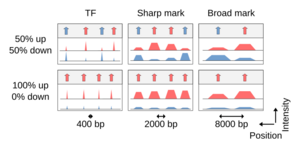
Name
sd_identity
Zweck
missing translation: trackingobject.sd_identity.desc
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
_li_dcdm_c
Zweck
missing translation: trackingobject._li_dcdm_c.desc
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
_gat_UA
Zweck
missing translation: trackingobject._gat_UA.desc
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
fc_session
Zweck
missing translation: trackingobject.fc_session.desc
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
__cf_bm
Zweck
Cloudflare Bot Manager, filtert Anfragen die vermutlich von Bots kommen.
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
_uetsid
Zweck
missing translation: trackingobject._uetsid.desc
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
_gid
Zweck
Wird verwendet, um Benutzer zu unterscheiden.
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
AF_SYNC
Zweck
missing translation: trackingobject.AF_SYNC.desc
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
_gcl_au
Zweck
Wird von Google AdSense zum Experimentieren mit Werbungseffizienz auf Webseiten verwendet.
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
_gcl_dc
Zweck
missing translation: trackingobject._gcl_dc.desc
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
_gcl_aw
Zweck
Google: Analytics, Advertising for Google Ads
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
_abexps
Zweck
Dieses Vimeo-Cookie hilft Vimeo, sich an die von Ihnen getroffenen Einstellungen zu erinnern. Dabei kann es sich zum Beispiel um eine voreingestellte Sprache, um eine Region oder einen Benutzernamen handeln. Im Allgemeinen speichert das Cookie Daten darüber, wie Sie Vimeo verwenden.
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
OptanonConsent
Zweck
Speichert Ihre Zustimmung zum Vimeo-Datenschutz.
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
OptanonAlertBoxClosed
Zweck
Speichert Ihre Zustimmung zum Vimeo-Datenschutz.
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
player
Zweck
Speichert die Einstellungspräferenzen (z.B. Lautstärke, Video Qualität, Untertitel)
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
_ttp
Zweck
missing translation: trackingobject._ttp.desc
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
_tt_enable_cookie
Zweck
missing translation: trackingobject._tt_enable_cookie.desc
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
_uetvid
Zweck
missing translation: trackingobject._uetvid.desc
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
_scid
Zweck
missing translation: trackingobject._scid.desc
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
_scid_r
Zweck
missing translation: trackingobject._scid_r.desc
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
_lc2_fpi
Zweck
missing translation: trackingobject._lc2_fpi.desc
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
sd_client_id
Zweck
missing translation: trackingobject.sd_client_id.desc
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
afUserId
Zweck
missing translation: trackingobject.afUserId.desc
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
_ga
Zweck
Wird verwendet, um Benutzer zu unterscheiden.
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
vuid
Zweck
Von Vimeo generierte ID zum erstellen von Statistiken für den Video Besitzer.
Ablauf
200 Monate
Typ
HTML
Anbieter
Vimeo
Name
_cfuvid
Zweck
Cloudflare cookie das zur begrenzung der übertragungsrate verwendet wird.
Ablauf
Session
Typ
HTML
Anbieter
Vimeo